Des modèles qui reconnaissent de mieux en mieux les plantes
⏱ 5 minDepuis 2011, le challenge PlantCLEF met en avant les progrès des modèles d’identification reposant sur l’apprentissage automatique appliqué à la botanique. L’arrivée des réseaux de neurones convolutionnels (CNN) a permis de nettes avancées. L’application Pl@ntNet met le dernier cri à la portée du plus grand nombre.
Identifier une plante peut être complexe, même pour un botaniste. L’idée d’utiliser des outils d’identification automatique à base de machine learning a été proposée dès 2004. Il existe aujourd’hui plusieurs applications d’identification de la flore à partir d’images prises avec un smartphone comme Seek, PlantSnap, PictureThis ou Pl@ntNet. Si vous avez coutume de les utiliser, vous aurez probablement constaté l’évolution de leurs performances. Et pour cause, les modèles de machine learning utilisés ainsi que les jeux de données d’entraînement ne cessent d’évoluer. Les chercheurs de Pl@ntNet ont pu suivre l’évolution de ces méthodes et de leurs performances en organisant le challenge d’identification de plantes PlantCLEF1. Retraçons l’histoire avec eux.
« Nous avons créé ce challenge en 2011, raconte Alexis Joly, chercheur Inria coresponsable du projet Pl@ntNet. Car Pl@ntNet est avant tout un projet de recherche théorique, initié en 2009, lauréat, fin 2020, du « Prix de l’innovation Inria – Académie des sciences – Dassault Systèmes« . Ce n’est qu’en 2015 que c’est devenu aussi une application grand public et une plateforme de sciences participative. » Téléchargée 20 millions de fois depuis 2013, elle est actuellement utilisée par 200 000 personnes chaque jour, et propose des identifications à partir de nombreuses flores régionales.
Son succès croissant tient aussi au réseau d’experts qui révisent les données lorsqu’elles sont proposées par les utilisateurs via un système de contribution. Ils nettoient ainsi la galerie d’images utilisée pour l’entraînement des modèles. Sans eux, les performances s’effondreraient progressivement… Depuis 2020, les observations les plus fiables sont également intégrées à la base de données mondiale sur la biodiversité du GBIF (Global Biodiversity Information Facility) où elles sont aussi révisées.
Une compétition annuelle
« Le challenge annuel PlantCLEF nous permet de comparer les approches, de suivre l’état de l’art, explique Alexis Joly. Selon les années, entre 5 et 15 équipes de recherche du monde entier y participent : elles travaillent plusieurs mois sur les jeux de données d’apprentissage que nous leur fournissons, tirées de Pl@ntNet. Nous évaluons ensuite les performances de la trentaine de modèles participants sur un jeu de données test. » En sept ans, les performances des meilleurs systèmes ont été considérablement améliorées (voir tableau ci-dessous). Le taux de reconnaissance des espèces est passé de 25 %, en 2011, à 92 % en 2017, sachant que le jeu de données d’apprentissage était de plus en plus fourni et complexe (plus d’un million d’images et 10 000 espèces en 2017 au lieu de 1 469 images et 71 espèces en 2011).
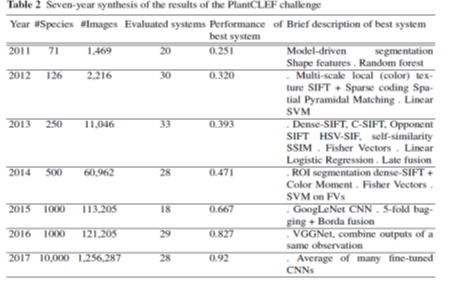
Que constate-t-on ? Qu’en 2011, les meilleurs résultats étaient obtenus avec des modèles à base de descripteurs de forme et des algorithmes de forêts aléatoires (random forest), que les performances s’améliorent au fil des ans en combinant plusieurs descripteurs (de couleur, de texture) et des algorithmes à base de machines à vecteurs de support (en anglais, support vector machine, SVM). Jusqu’à la première utilisation du deep learning et des réseaux de neurones convolutionnels (en anglais convolutionnal neural networks, CNN) en 2015. Aujourd’hui, ce sont de nouvelles architectures, les « transformers », qui entrent en compétition.
Les meilleures solutions intégrées dans Pl@ntNet
« À la lumière de ces résultats, nous faisons évoluer l’application Pl@ntNet qui a ainsi connu un véritable saut qualitatif en 2015, quand nous avons remplacé les six algorithmes à base de descripteurs par un seul réseau de neurones convolutifs, précise le chercheur2. Nous menons également nos propres tests sur la totalité des données de Pl@ntNet. » Mais cela ne suffit pas. L’application comporte par ailleurs un mécanisme de filtrage géographique par grandes régions (un filtrage pas trop exclusif car les plantes sont plus mobiles qu’on ne pense), ainsi qu’un moteur de recherche d’images similaires (baptisé Snoop), qui permet de présenter à l’utilisateur une photo de la plante la plus probable mais aussi d’autres qui lui ressemblent pour corriger l’identification si besoin. Pour que les données contributives permettent d’améliorer l’algorithme, un mécanisme accorde plus ou moins de crédit à chaque correction selon le profil de compétences attribué à l’utilisateur3.
La recherche du meilleur compromis réserve des surprises. « Par exemple, les identifications de Pl@ntNet ont été considérablement améliorées lorsque nous avons intégré des données issues du web dans l’entraînement de notre réseau de neurones convolutifs, souligne Alexis Joly. Ainsi, contre toute attente, l’apprentissage est moins efficace à partir d’images validées par des experts qu’à partir d’immenses quantités de données bruitées (non révisables car elles sont sous licence, nous ne pouvons pas les intégrer à notre galerie de photos que les experts ou les utilisateurs révisent)4. »
« Autre surprise, poursuit le chercheur : il s’avère que le meilleur modèle de CNN pour Pl@ntNet est actuellement Inception v3 (de Google) et non l’un de ces modèles issus d’approches d’optimisation d’architecture neuronale (Neural Architecture Optimization, NAO) qui occupent le devant de la scène de challenges réputés comme ImageNet [la référence en matière de concours de reconnaissance visuelle d’objets par intelligence artificielle, NDLR]. » Les chercheurs ne savent pas encore expliquer ces résultats. Si ce n’est que les problématiques d’identification d’images ne sont pas du même ordre : Pl@ntNet analyse des détails très fins sur 35 000 classes de plantes aujourd’hui (la flore mondiale est évaluée à 400 000 espèces) alors qu’ImageNet ne comporte que 1 000 classes.
Un outil pour amateurs… apprécié par les pros
Outre le grand public qui apprend à reconnaître telle ou telle plante, Pl@ntNet est devenu un outil prisé des botanistes : l’application leur sert de pense-bête et leur permet par exemple de détecter des espèces qui portent plusieurs noms selon la flore de référence utilisée, et de proposer des solutions.
L’identification automatique de plantes soulève également d’intéressantes questions d’analyse de données. « Un de nos doctorants travaille sur la gestion de l’incertitude dans le cas de plantes avec des fleurs similaires, explique Alexis Joly. Cela nous permettra bientôt de proposer plusieurs espèces de manière certaine plutôt qu’une liste ordonnée d’espèces. Autre exemple, les recherches menées avec l’Imag (Institut montpelliérain Alexander-Grothendieck) sur ce déséquilibre qui caractérise notre jeu de données : pour quelques espèces, nos algorithmes sont entraînés sur un très grand nombre d’images alors qu’une centaine de milliers d’espèces ne sont représentées que par quelques images. Pourquoi notre CNN converge-t-il malgré cela ? Y a-t-il un transfert de connaissances d’une espèce à l’autre ? »
Isabelle Bellin
Notes :
1. A. Joly, H. Goëau, H. Glotin, C. Spampinato, P. Bonnet, W.P. Vellinga, … & H. Müller, “Biodiversity Information Retrieval through Large Scale Content-based Identification: a Long-Term Evaluation”, In Information Retrieval Evaluation in a Changing World, 2019, Springer. ⟨hal-02273280⟩
2. A. Affouard, H. Goëau, P. Bonnet, J.C. Lombardo & A. Joly, “Pl@ntnet App in The Era of Deep Learning. In ICLR: International Conference on Learning Representations“, 2017. ⟨hal-01629195⟩
3. M. Servajean, A. Joly, D. Shasha, J. Champ & E. Pacitti, “Crowdsourcing Thousands of Specialized Labels: a Bayesian Active Training Approach“, in IEEE Transactions on Multimedia, 2017,19(6). ⟨hal-01629149⟩
4. H. Goëau, P. Bonnet & A. Joly, “Plant Identification Based on Noisy Web Data: The Amazing Performance of Deep Learning (LifeCLEF 2017)“, in CLEF: Conference and Labs of the Evaluation Forum, 2017. ⟨hal-01629183⟩
Image de une : © Pl@ntNet